使用卷积神经网络,实现无肿瘤分割的全脑MRI胶质瘤生存预测
传统上,成人弥漫性胶质瘤在组织病理学上分为世界卫生组织(WHO)II级、III级(低级别胶质瘤,LrGG)和IV级(胶质母细胞瘤,GBM)。
弥漫性胶质瘤是成人最常见的颅内原发性恶性肿瘤之一,复发率极高。传统上,成人弥漫性胶质瘤在组织病理学上分为世界卫生组织(WHO)II级、III级(低级别胶质瘤,LrGG)和IV级(胶质母细胞瘤,GBM)。2016年和2021年的最新WHO标准对分类进行了调整,强调了分子标志物如异柠檬酸脱氢酶(IDH)突变等新的指标以实现更准确的分类。
最近,建立在大型标记医学图像数据库上的卷积神经网络(CNN)在疾病分类任务中显示出巨大潜力。然而与诊断相比,CNN的预后性能还没有得到全面的研究。另一方面,CNN需要大量的标记图像进行模型训练才能达到可接受的准确性。为了从CT或MRI中训练出一个准确的模型,大多数CNN需要分割的病变图像作为输入。然而,手动分割MRI中的胶质瘤是一项耗时的工作,自动化的肿瘤分割算法也需要手动分割的肿瘤图像作为训练实例。因此,建立一个基于CNN的免去肿瘤分割的生存预测模型,无疑具有巨大的临床效益。
近日,发表在European Radiology杂志的一项研究开发和验证了一个用于预测术前全脑MRI的胶质瘤总生存率且无需对肿瘤进行分割的深度CNN(DeepRisk),并将DeepRisk与根据专家分割的肿瘤图像建立的传统深度学习模型进行了比较,同时评估了DeepRisk在临床和基因图谱中的预后价值。
本项多中心回顾性研究建立了两个深度学习模型用于从MRI中预测生存率,包括从全脑MRI中建立的DeepRisk模型以及从专家分割的肿瘤图像中建立的原始ResNet模型。两个模型都是使用训练数据集(n = 935)和内部调整数据集(n = 156)开发的,并在两个外部测试数据集(n = 194和150)和TCIA数据集(n = 121)上进行测试。C-指数、综合Brier得分(IBS)、预测误差曲线和校准曲线被用来评估模型的性能。
总共有1556名患者入选(年龄,49.0±13.1岁;830名男性)。DeepRisk评分是一个独立的预测因素,可以将每个测试数据集中的患者分层为三个风险亚组。DeepRisk的IBS和C-index在外部测试数据集1中分别为0.14和0.83,在外部数据集2中为0.15和0.80,在TCIA数据集中为0.16和0.77,这与原始ResNet的预测结果相当。DeepRisk在6、12、24、26和48个月的AUC在0.77和0.94之间。将DeepRisk评分与临床分子因素结合起来,得到的提名图比临床列线图具有更好的校准和分类准确性(净分类改善0.69,P < 0.001)。
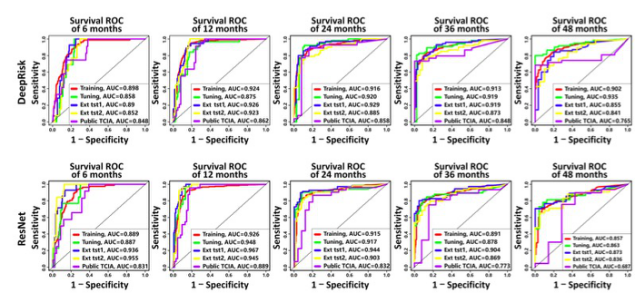
图DeepRisk(上行)和ResNet(下行)模型在6、12、24、36和48个月时分别在训练数据集(红色)、调整数据集(绿色)、外部测试数据集1(蓝色)和2(黄色)以及公共TCIA数据集(紫色)上的随时间而变化的ROC曲线
本研究所提出的深度学习模型可直接从术前全脑MRI中预测胶质瘤的总生存率,且不需要进行肿瘤分割,其性能与使用精确分割的肿瘤图像建立的ResNet模型相当。该模型的输出可进行患者的术前风险分层评分,且与现有的临床因素和IDH突变状态相比具有独立的预后价值。
原文出处:
Zhi-Cheng Li,Jing Yan,Shenghai Zhang,et al.Glioma survival prediction from whole-brain MRI without tumor segmentation using deep attention network: a multicenter study.DOI:10.1007/s00330-022-08640-7






